Ahoj, já jsem Ondra a toto je moje rubrika IT bravíčko
TMHMM je algoritmus, který na základě proteinových sekvencí aminokyselin predikuje přítomnost transmembránových segmentů. To jsou ty části peptidického řetězce proteinu, které procházejí biologickými membránami. Základní idea je taková, že aminokyselinová sekvcence transmembránových proteinů je biologicky naprogramovaná a evolucí vytříbená, aby v ní byly jasně ohraničené úseky, které „patří“ do biomembrán.
(Nutno podotknout, že aby byl protein pak zasazený do membrány, tak ho tam musí během jeho syntézy (translace) aktivně cpát, a takový mechanismus skutečně existuje, nicméně s sebou nese další nutnou přítomnost biologického kódu, a to takového, který umožňuje rozpoznat, na které straně membrány má být která část daného proteinu. Tato problematika je pro buňku logisticky mnohem zásadnější čurbes, než samotná existence transmembránových úseků, ale to sem nyní nepatří.)
Tyto úseky se primárně vyznačují tím, že v nich jsou přítomné preferenčně hydrofobní (ve vodě „nerozpustné“) aminokyseliny, ale spíš některé z nich (některé na to nejsou moc vhodné, jsou třeba trochu moc „velké“), úsek bývá do jisté míry ohraničen, například pozitivně nabitými aminokyselinami (protože biomembrány mají tentenci mít záporně nabité polární „okraje“) atd.
Každopádně TMHMM je tu s námi přes 23 let a trochu zastaral. Fungoval jako webová aplikace, ostatně je stále k dispozici. Před dvěma lety byla uvedena nová lepší (předpokládám :)) verze DeepTMHMM. Minimálně je lepší v tom, že jasně rozpozná tzv. signální sekvenci, tedy část proteinu, který je sice de facto transmembránovým úsekem, ale pro účely nejčastěji sekrece proteinů do mimobuněčného prostoru bývá odštěpen a degradován.
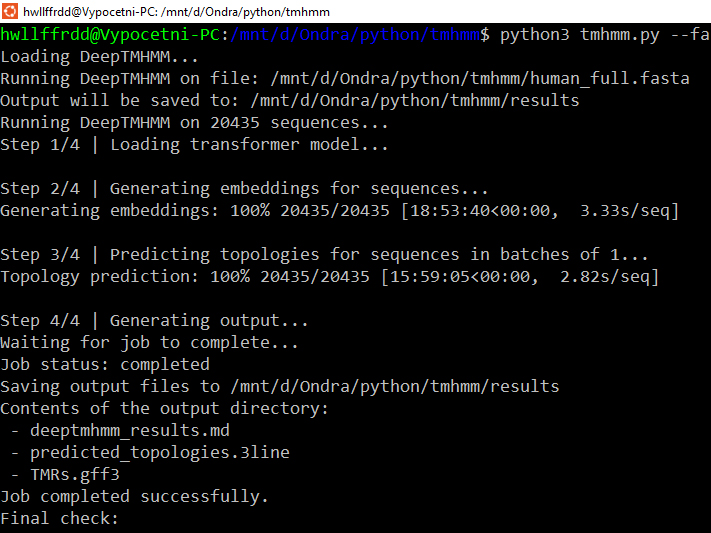
Doba je zlá, a časy, kdy jste si mohli nacpat 20 MB proteinových sekvecí do analýzy a pustit ji na serveru autorů aplikace, jsou pryč (ale fungovalo to tak skutečně dlouho!). Pokud někdo skutečně potřebuje nacpat do analýzy 20 MB proteinových sekvecí (já), může buď soubory serveru kouskovat, a cpát je tam postupně po dobu několika týdnů, a nebo, pokud si troufne, napsat skript v Pythonu, a po dobu několika týdnů se s ním trápit, než ho zprovozní (studiový smích).
Ano, to přesně se mi stalo, reálně si velmi vážím dokumentace, kterou autoři připravili, ale tak řekněme, je to prostě dokumentace, teoreticky nad tím máte pochopit co s tím, ale... um... no, chápete. Reálně jsem měl se skriptem dva zásadní problémy. Zaprvé, když jsem to pouštěl v PyCharmu ve Windows, neřešitelným problémem bylo uložení byť jen dočasných souborů na disk. Kamkoli na disk. Nešlo to prostě. Nakonec pomohlo nainstalovat Ubuntu, nakonfigurovat WSL2 a připojit k němu Docker.
Pak přišel druhý zásek, skript doběhl a neuložil žádné soubory. Pokud kouknete na zmíněnou dokumentaci jistě tušíte, že chyba je v tomto případě mezi židlí a klávesnicí, protože v dokumentaci je jasně popsané, jaké příkazy dodat, aby se soubory uložily. Nakonec mě ale musil popostrčit jeden z autorů, když jsem se zoufale ptal na jejich Slacku, co s tím.
No nic finální kód je zde, vytěžená data z lidského proteomu jsou použita ve webové aplikace Human IMPs a já budu (možná) víc věřit v dokumentace (snad) (těžko říct).